林茹,李可萌,刘娟娟,戴剑漉,赫卫清
·论著·
螺旋霉素生物合成基因簇中,-二甲基转移酶基因功能的鉴定
林茹,李可萌,刘娟娟,戴剑漉,赫卫清
100050 北京,中国医学科学院北京协和医学院医药生物技术研究所卫健委抗生素生物工程重点实验室/医科院合成生物学重点实验室
确定螺旋霉素(SP)生物合成途径中编码两个,-二甲基转移酶的基因9 和22 的功能,并在基因阻断变株中分离纯化相应的 SP 前体物。利用抗性基因插入或 CRISPR-Cas 基因编辑的方法阻断9 和22 基因,并对其进行基因回复实验。利用薄层色谱(TLC)和高效液相色谱-质谱(HPLC-MS)联用分析变株 Δ9 和 Δ22的发酵产物。利用半制备液相色谱的方法分离纯化缺少甲基化修饰的 SP 前体物,经核磁共振波谱(NMR)数据分析确定其化学结构。成功构建了两个基因的阻断株 Δ9 和 Δ22。Δ9 变株的发酵液没有抗菌活性的物质产生,经 HPLC-MS 分析也未发现相应的 SP 前体物。而 Δ22 变株的发酵产物的抗菌活性下降 50% 以上,从 Δ22 变株发酵产物中分离出福洛胺糖缺少两个甲基的 SP 前体物。22 基因负责 SP 生物合成中福洛胺糖上的,-二甲基的催化,而9基因可能参与麦洛胺糖的,-二甲基化修饰,且两者在功能上不能互补。
螺旋霉素;
,-二甲基转移酶基因;
基因阻断
螺旋霉素(spiramycin,SP)是一种大环内酯类抗生素(macrolide antibiotics,MA),它的主核是一个16 元内酯环,其上连有 3 个脱氧糖,分别是 2 个氨基糖——福洛胺糖、麦洛胺糖和 1 个中性糖——麦洛糖,这些糖基是 SP 抗菌活性的关键基团(图 1)。MA 属细菌生长期速效抑菌剂,一般无过敏性反应,口服吸收好,服用方便。许多半合成改造后的 MA 在胃肠道中的稳定性好,组织分布广,体内持续时间长,但生物利用率较低,胃肠道反应比较大。然而,16 元环的 SP 没有其他 MA 所致的肝损害,对胃肠道的反应也小于 14 元环 MA[1-5]。MA 是通过与细菌 50S 核糖体结合而抑制其蛋白质合成。与 14 元环相比,16 元内酯环上多了 2 个碳原子、C3 位没有糖基、C5 上有两个糖基,特别是 SP 在 C9 位有一个福洛胺糖,这些结构上的差异导致其与核糖体大亚基的结合位点也不同。C5 位较长的糖基侧链能干扰到蛋白合成中的肽酰基转移酶反应,最终引起抑制活性不同。此外,药物在组织中的渗透性对于药物的作用十分重要。螺旋霉素类药物在体内能迅速而广泛地分布至组织,大部分组织的浓度高于血药浓度 50 倍以上[6];
除此之外,该类药物能很好地渗透入细胞内发挥作用,在体内能提高吞噬细胞的吞噬功能和持续的抗生素后效应,且安全性好[7-8]。国家一类新药可利霉素[9]的成功研制与选择 SP 这个安全的母核有很大关系。
SP 的生物合成已在生二素链霉菌()ATCC 23877 中进行了深入研究,现已完成此株菌的全基因组测序[10],对 SP 生物合成基因簇组成[11]、调控基因[12-13]、糖基转移酶基因[14]和后修饰基因[15]已经进行深入研究和报道,基本阐明了 SP 生物合成的主要过程。但糖基的生物合成基因功能只从生物信息学上进行了推测,未能进行实验确证。本实验室采用的SP 产生菌是从我国甘肃永昌县土壤中分离得到的,其形态及培养特征与国外报道的SP 产生菌相比,有明显的不同;
与其他文献报道的近似种也不尽相同,命名为螺旋霉素链霉菌[16]。从中也鉴定出了SP 生物合成基因簇(NCBI 基因登录号MH460451,基因命名以开头),相关的基因排列见图 1。根据生物信息学分析推测出 42 个orf,与ATCC 23877 中公布的基因排列基本相似,序列的同源性在 70% 左右,但也有一些基因存在差异,如ATCC 23877 中没有2、20的同源基因。其中与 SP 的糖基合成相关基因包括9、16 ~ 22、24、26、28、29、33 ~ 35 和 41,共16 个基因,糖基转移相关基因7、8、30 ~ 32、40 等6 个,其中通过同源性推测:7 编码麦洛胺糖的糖基转移酶;
8 编码其糖基化辅助蛋白;
31 编码福洛胺糖的糖基转移酶;
30 编码其糖基化辅助蛋白;
40 编码麦洛糖的糖基转移酶。
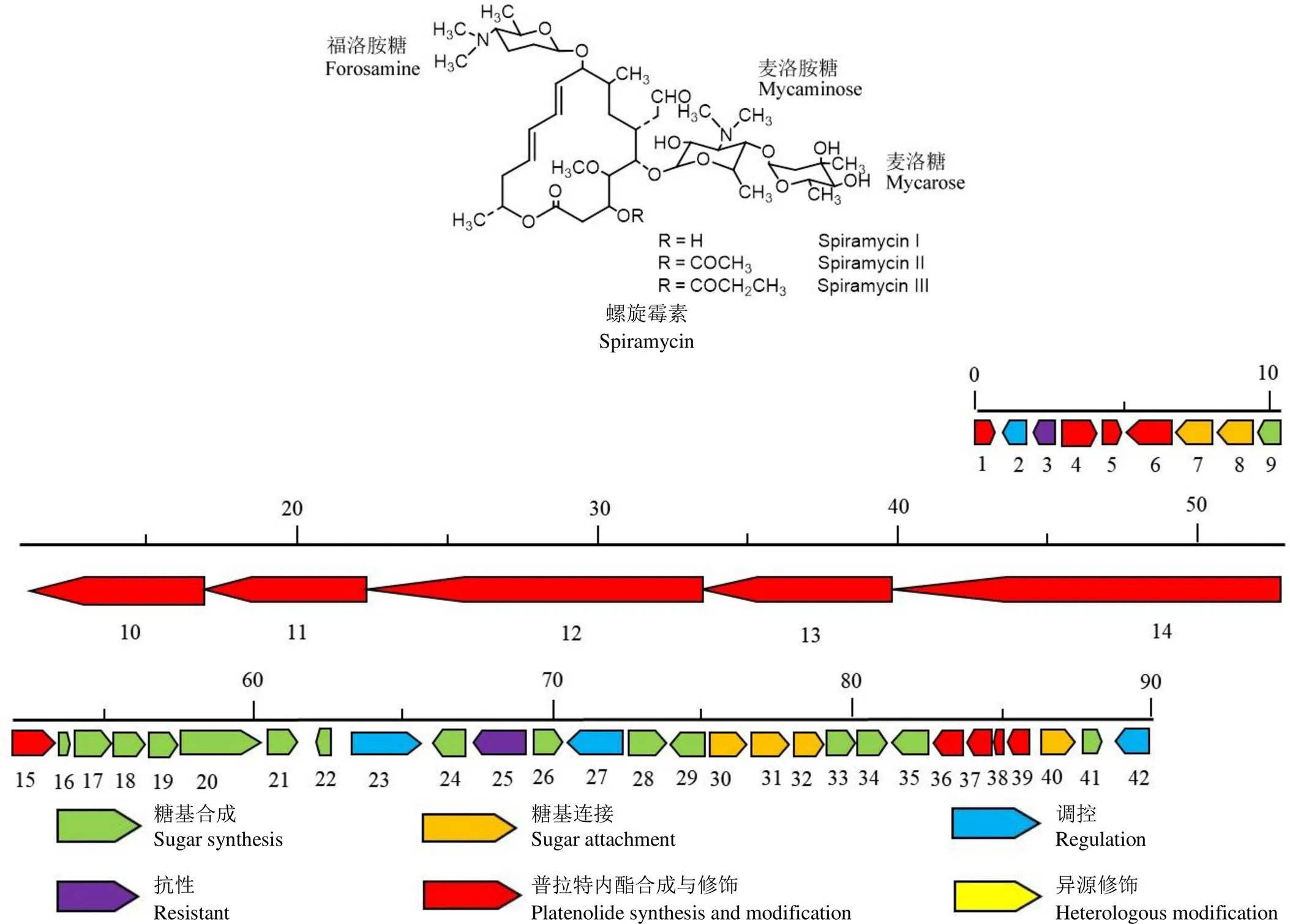
图1 螺旋霉素及其生物合成基因簇
Figure 1 Structure of spiramycin and genetic organization of the spiramycin biosynthetic gene cluster
SP 的两个氨基糖上的二甲氨基是抗菌活性的重要基团,将其去掉就会严重影响 SP 的抗菌活性[17-18]。通过生物信息学推测,在 SP 基因簇中9和22为,-二甲基转移酶基因,但并没有文献报道哪个基因负责相应氨基糖的二甲基化修饰。本文将对9和22基因分别进行阻断和回复实验,并从基因阻断株中分离缺少二甲基修饰的 SP 前体物来鉴定两个基因的具体功能。
1.1 材料
1.1.1 菌种及质粒 SP 产生菌1941 由本实验室保存;
大肠杆菌BL21 (DE3) 及DH5α 感受态细胞均购自北京全式金生物技术有限公司;
枯草芽孢杆菌CPCC 100029 为本实验室保存。
pGH112 载体[19]是一种在链霉菌中不稳定遗传的载体,带有硫链丝菌素抗性基因。pUWL201PW 大肠杆菌和链霉菌的穿梭载体为本实验室保藏;
CRISPR-Cas9 基因编辑质粒 pKCcas9do[20]由中国科学院上海生命科学研究院姜卫红研究员惠赠;
本实验中所涉及引物见表 1。
1.1.2 试剂 氨苄青霉素(ampicillin,Amp)、安普霉素(apramycin,Apr)和硫链丝菌素(thiostrpton,Thio)均购自美国 Sigma-Aldrich 公司;
SP 购自河南天方药业股份有限公司;
限制性内切酶及 T4 连接酶均购自日本 Takara 公司;
色谱级乙腈和甲醇购自北京市通广精细化工公司。
表1 本实验所用引物
Table 1 Primers used in this study
寡核苷酸Oligos序列(5"-3")Sequence (5"-3")功能Use bsm22-LFCCGGAATTCCACATCGAGCAGGCCGTCAC (EcoR I)用于构建阻断bsm22基因的质粒pGH-bsm22Construction of pGH-bsm22 plasmid for inactivationof the bsm22 gene bsm22-LRAACTGCAGGCAAGAAGACCCGGATGGAAAT (Pst I) bsm22-RFCGCGGATCCAGCCGTCCAGGAAGGTGTCG (BamH I) bsm22-RRCTAGTCTAGAAAGCCAGCGGGCAGATATGAA (Xba I) bsm22-IFACGAGGTGACCGACAGCGACTTΔbsm22 突变株鉴定引物Identification primer for Δbsm22 mutant bsm22-IRGACCAGGCTGCCGTTCCTT bsm22-CFGTGGGGGCCTGGGGAGTG用于Δbsm22 突变株的回复Complementation of Δbsm22 bsm22-CRACCGCAGCGTGGGCATCG bsm9-gDNA-1GGACTAGTAGAATGCGAGACCCGGCAGAGTTTTAGAGCTAGAAAT (Spe I)用于构建阻断bsm9 基因的质粒pKCas-bsm9Construction of pKCas-bsm9 plasmid for inactivationof the bsm9 gene bsm9-gDNA-2GGACTAGTCGGGGTTGCGGTCGGTCGCGGTTTTAGAGCTAGAAAT (Spe I) bsm9-downCGGAATTCCTCAAAAAAAGCACCGACTCGG (EcoR I) bsm9-LFCGGAATTCCACTCCGAGGAACCCGAGC (EcoR I) bsm9-LRGGACTGTTCATCGGCACCC bsm9-RFGGCGGGTGCCGATGAACAGTCCCAGCATGTCGTGGGAGAGCT bsm9-RRCCCAAGCTTGAGTACGTCCCCTTCGCCG (HindIII) bsm9-IFGCGGGTGCCGATGAACAGΔbsm9 突变株鉴定引物Identification primer for Δbsm9 mutant bsm9-IRAGGGGAAGGGCAAGGACTAC bsm9-CFGGAATTCCATATGTACGCCAACGACATCGCGG用于Δbsm9 突变株的回复Complementation of Δbsm9 bsm9-CRCCCAAGCTTTCAGCGGCGGGTGCCGAT
1.2 方法
1.2.122 基因阻断株的构建
1.2.1.122 阻断质粒的构建 以1941 基因组 DNA 作为模板进行PCR,分别利用两对引物22-LF/22-LR 和22-RF/22-RR 扩增22 基因两端的左右同源臂。用R I 和I 酶切后得到 1105 bp 的左同源臂,用H I 和I 酶切后得到 1056 bp 的右同源臂,同时利用I 和H I 酶切 pUC19-(3)IV 重组质粒得到 Apr抗性基因(3)IV,将上述 3 个基因片段与经R I 和I 酶切后的线性化质粒 pGH112 进行连接得到重组质粒 pGH112-22::(3)IV。
1.2.1.222基因阻断菌株的筛选 将重组质粒 pGH112-22::(3)IV 转化至ET12567/pUZ8002 感受态细胞中,通过原生质体转化的方式转入到1941。根据同源双交换插入失活的原理构建1941 的22 的阻断株(Δ22)。pGH112-22::(3)IV属于不稳定质粒,经过多次传代培养后质粒会丢失,发生同源双交换的突变菌株表现为 ThioSAprR表型。利用含Thio 和 Apr 抗性平板筛选转化子:首先将转化子转接至含有 50 μg/ml Apr 的斜面培养基培养,通过在无抗性平板上多次传代,筛选出表型为ThioSAprR的突变株。再通过 PCR 验证,最终获得正确突变株 Δ22。
1.2.29 基因阻断株的构建
1.2.2.19 阻断质粒的构建 以1941 基因组 DNA 作为模板,利用引物9-LF/LR 和9-RF/RR对9基因敲除片段的左右同源臂进行扩增。将两段 DNA 片段切胶回收后,利用重叠延伸 PCR,以9-LF 和9-RR 为引物扩增完整的9 基因改造片段,并用R I 和d III 进行酶切。
分别利用引物91、92 与9-down 配对,以pKCcas9do 质粒为模板进行 PCR,获得两条 100 bp 左右的引导 DNA(guide DNA,gDNA)序列 gDNA-1 和 gDNA-2,用I和R I 对其进行酶切。再分别将 gDNA-1、gDNA-2 与9 基因改造片段克隆至 pKCcas9do质粒的I 和d III 位点获得重组质粒 pKCas-Δ9-1 和 pKCas-Δ9-2。
1.2.2.2基因阻断株的筛选 将构建好的重组质粒 pKCas-Δ9-1 和 pKCas-Δ9-2 通过原生质体转化法分别导入到1941 中,1 μg/ml Thio 诱导 Cas9 表达,利用gDNA 的转录产物 gRNA 引导 Cas9 切割9 基因的靶位点,形成双链断裂,重组质粒上带有同源臂的9 基因敲除片段通过同源双交换整合至靶位点。将目的菌株于 37 ℃培养,使质粒停止复制,通过传代获得质粒丢失的目标菌株。利用引物9-IF 和-IR 进行 PCR 筛选获得正确的9基因阻断株(Δ9)进行验证。
1.2.3 Δ9 和 Δ22 回复株的构建 以1941 基因组DNA 为模板扩增出完整的9 和22 基因,克隆在 pUWL201PW 载体的组成型启动子的下游,获得回复用的质粒 pUW-9 和 pUW-22,将其分别转化至制备好的突变株 Δ9 和 Δ22 原生质体内,用含Thio 抗性的平板筛选转化子,利用菌落PCR 鉴定出正确的9 和22 基因回复菌株。
1.2.4 菌株发酵 将变株 Δ9、Δ22 及.1941 菌株分别接种于斜面培养基,28 ℃传代培养 7 ~ 10 d,挖取 1 cm2菌块接种到装有 100 ml 的发酵培养基的三角瓶中,于 28 ℃、200 r/min 振荡培养 5 d,发酵液 10 000 r/min 离心 10 min,收集上清液。
1.2.5 发酵产物的抗菌活性检测及提取 吸取离心后的上清液 200 μl,用牛津杯法测定发酵产物抗枯草芽孢杆菌的活性。剩余上清液用 5 mol/L NaOH 调节 pH 至 8.5 ~ 9.0 后,用等量的乙酸乙酯萃取 8 h 以上。吸取萃取液,于通风橱吹干后溶于 100 μl乙酸乙酯或甲醇,备用。
1.2.6 发酵产物的 TLC 及 HPLC 检测
1.2.6.1 TLC 检测 挥干后的发酵液溶于 100 μl 乙酸乙酯,于薄层硅胶板初步分析。展开体系为乙酸乙酯:环己烷:甲醇:氨水= 6:4:1:0.2(/),展开后将硅胶板于 254 nm 下观察,并用碘熏显色。比较变株 Δ9、Δ22 及.1941 菌株的发酵产物。
1.2.6.2 HPLC 分析 挥干后的发酵液经甲醇复溶后,0.22 μm 滤膜过滤,取 6 μl 进行 HPLC 检测。采用安捷伦高效液相色谱仪1200 Series、YMC-Triart C18 柱(250 mm × 4.6 mm,5 μm),流动相为乙腈/10 mmol/L pH 8.0 醋酸铵(60/40),等度洗脱 30 min,检测波长231 nm,流速1 ml/min。以.1941 发酵粗提物为对照组,通过比较保留时间与紫外吸收初步判定变株 Δ9 和 Δ22是否有目标产物生成。
1.2.6.3 LC-MS 分析 LC-MS 检测条件参照1.2.6.2,采用美国Thermo 公司 LTQ 型液相色谱-质谱联用仪检测。
1.2.7 目标产物的分离纯化
1.2.7.1 硅胶柱层析 发酵约 10 L Δ22 菌株,经乙酸乙酯萃取并旋干浓缩后加入 1.5 倍硅胶粉,干法上样进行硅胶柱层析。用三氯甲烷:甲醇:氨水(10:1:0.2)进行梯度洗脱,分步收集并检测,将所需部分合并旋蒸,于–20 ℃ 冻存。
1.2.7.2 凝胶柱层析 经硅胶柱层析后得到的样品用少量乙腈溶解并用 0.45 μm 滤膜过滤,利用制备液相制备。色谱柱:YMC-Pack ODS-A(250 mm × 20 mm,5 μm),流动相:A 相为水(色谱级氨水调至 pH 8.0),B 相为乙腈,30% ~ 100% 梯度洗脱50 min。检测波长:231 nm,流速:5 ml/min。收集目标产物,冷凝蒸干。
1.2.7.3 半制备液相纯化 少量乙腈溶解样品,0.22 μm 滤膜过滤,经岛津 LC-20A 高效液相色谱仪进行半制备。色谱柱:ReproSil-Pur Basic C18(250 mm × 10 mm,5 μm),流动相:乙腈/水(色谱级氨水调至 pH 8.0)(55/45),等度洗脱 30 min。检测波长231 nm,流速2.5 ml/min。收集目标产物,旋转蒸干再冷冻干燥,待完全除去有机试剂和水后称重。
1.2.7.4 NMR 分析 制备的样品(干粉)约10 mg 送中国医学科学院药物研究所测定1H 和13C 图谱,溶剂为 CD3OD-4。
2.1 bsm9及 bsm22基因的生物信息学分析
两个氨基糖的生物合成途径推测如图 2,参与福洛胺糖生物合成基因的按催化顺序包括:18、19、20、29、2621 和22(或9),参与麦洛胺糖生物合成基因按催化顺序包括:18、19、16、17 和22(或9)。其中9 和22 具体负责哪个氨基糖的二甲基化修饰还没有确定。经生物信息学推测,9 大小为 714 bp,编码 237 个氨基酸,上游与编码聚酮合酶的10 基因是在同一个转录单元中,因此为了不影响10 基因的转录与翻译,只能对9 基因中间的 orf 进行删除;
而其下游基因为糖基转移酶辅助蛋白基因8,有相应的启动子区,9 基因的缺失不影响其进行转录。22基因大小为720 bp,编码 239 个氨基酸,有自己的启动子和终止子,因此对其进行抗性基因插入失活,通常不会影响邻近基因的表达。虽然Bsm9 和Bsm22 都是,-二甲基转移酶,但序列一致性为 52%,说明两者之间存在较大差异。

图2 福洛胺糖和麦洛胺糖的生物合成途径[11]
Figure 2 The biosynthetic pathways of forosamine and mycaminose[11]
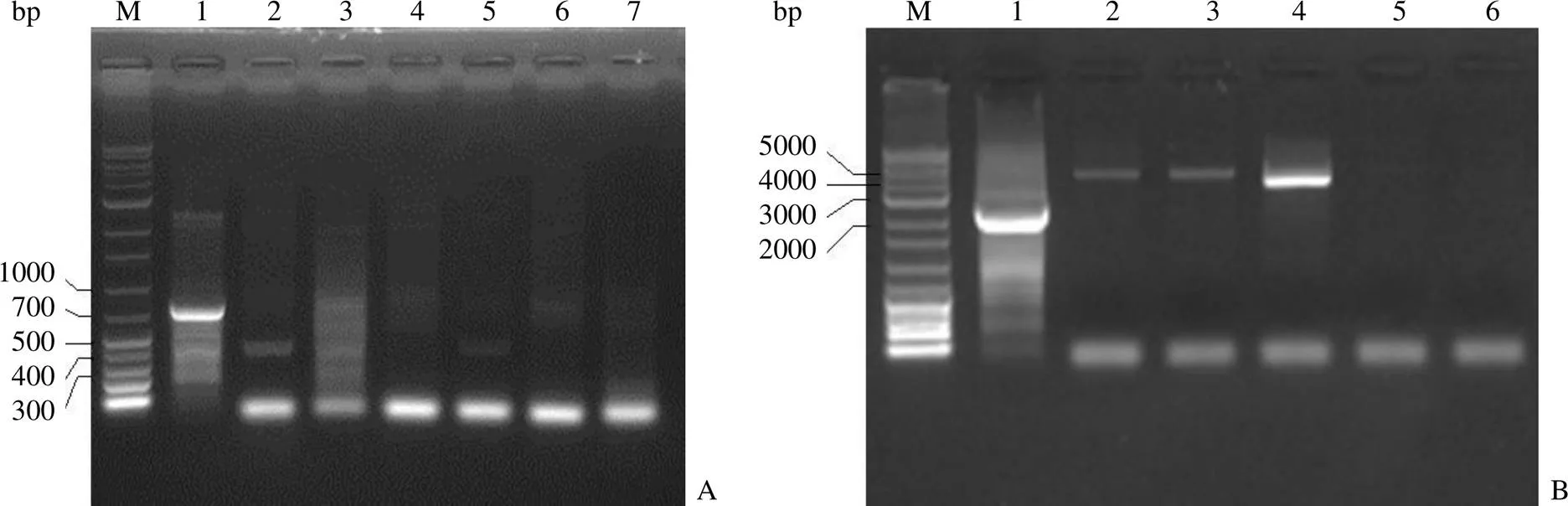
M:1 kb ladder marker;
1:野生型;
2 ~ 7:突变株
Figure 3 Identification of Δm9 (A) andΔ22 (B) mutants by PCR
2.2 bsm9 和 bsm22阻断株鉴定
首先对 Δ9 进行 PCR 鉴定,利用鉴定引物9-IF/IR 从原株基因组 DNA 中克隆出约 700 bp 的原始片段,变株中的序列应该删除了 300 bp,预期片段大小为 400 bp 左右。PCR 验证结果如图 3A 所示,第 2 和 5 泳道的变株为阳性克隆,扩增出的片段大小约 400 bp,将其进行测序后证实与设计的9 基因删除序列完全一致,说明获得了正确的9 基因失活突变株。利用鉴定引物22-IF/IR 从原株基因组 DNA 中克隆出 2.4 kb 左右的片段,而在 Δ22 变株中扩增出了插入 Apr 抗性基因片段后的大小为 4.2 kb左右(图 3B),通过测序最终证实得到了正确的22 基因的抗性基因插入失活株 Δ22。通过两个基因的回复引物9-CF/CR 和22-CF/CR 也证实完整的9 和22 基因又分别导入了 Δ9 和 Δ22 变株中获得了相应的基因回复株。
2.3 Δbsm9 和 Δbsm22 发酵产物的检测
对 Δ9 和 Δ22 基因阻断株以及两者的回复株分别进行发酵,并对发酵产物进行 TLC 检测,如图 4A 所示,与野生型1941 相比,Δ9 变株的发酵产物失去了抗菌活性,其产物在 TLC 展开并碘熏后未出现 SP 的三种组分,而 Δ9 的回复株又恢复了 SP 的合成。Δ9 发酵产物经过 HPLC-MS 分析也未发现与SP 及其前体物相关分子量的化合物。而 Δ22 变株的产物还保留一定的抗菌活性(图 4B),TLC 分析也发现其产物中含有部分与 SP 类似的化合物,Δ22 的回复株也可以正常产生 SP 的三个组分。

图4 Δbsm9 和 Δbsm22 变株发酵产物的鉴定[A:Δbsm9 发酵液抑菌活性和 TLC 分析(1:SP;
2:S. spiramyceticus 1941(野生型)发酵产物;
3:Δbsm9 发酵产物;
4:Δbsm9 回复菌株发酵产物);
B:Δbsm22 发酵液抑菌活性和 TLC 分析<(1:SP;
2:S. spiramyceticus 1941(野生型)发酵产物;
3:Δbsm22 回复菌株发酵产物;
4:Δbsm22 发酵产物)]/p>
Figure 4 The analysis of the fermentation products in Δ9and Δ22 disruption mutants and complementary mutants [A: Antimirobial activity of Δ9fermentation broth and TLC analysis of Δ9fermentation broth extraction (1: Spiramycin;2:1941 (wild type); 3: Δ9; 4:Δ9 complementation strain); B: Antimirobial activity of Δ22fermentation broth and TLC analysis of Δ22fermentation broth extraction (1: Spiramycin; 2:1941 (wild type); 3: Δ22 complementation strain; 4: Δ22)]

图5 Δbsm22 变株及回复株发酵产物的 HPLC-MS 检测[A:S. spiramyceticus 1941(野生型)发酵产物;
B:Δbsm22 发酵产物;
C:Δbsm22 回复菌株发酵产物;
D:SP I;
E:SP II;
F:SP III]
Figure 5 HPLC-MS analysis of Δ22 and its complementary strain fermentation broth [A:1941 (wild type);B: Δ22; C: Δ22complementation strain; D: SP I; E: SP II; F: SP III]
对 Δ22 变株的发酵产物进行 HPLC-MS 分析,从图 5B 中可以发现Δ22 变株发酵产物中检测到与 SP 保留时间相似的特征峰,它们的保留时间比野生型1941 的发酵产物峰SP I、SP II、SP III 略微提前,紫外吸收与对应的 SP I、SP II、SP III 几乎一致。进一步通过 LC-MS 鉴定,质谱显示化合物 I、II、III 的准分子离子峰分别是 816.4 [M+H]+、858.5 [M+H]+、872.5 [M+H]+,与 SP 缺失两个甲基的分子量一致,推测发酵产物可能为 SP 缺少甲基的前体物。根据上述质谱数据,推测这些可能的前体物 I 的分子式 C41H70N2O14,II 分子式为 C43H72N2O15,III 分子式为 C44H74N2O15。
表2 化合物II 的核磁波谱分析
Table 2 NMR of compoundII in CD3OD

2.4 Δbsm22产物的结构鉴定
因为化合物 II 的产量比较高,因此通过半制备液相分离了化合物 II,纯化后的化合物 II 为淡黄色粉末,之后进行1H和13C 核磁波谱数据分析(表2),1H-NMR(600 MHz,CD3OD-4)谱在2.5 ppm 附近存在多个甲基信号峰,与文献报道中 SP 的 7"和 8" 甲基的化学位移一致[21]。此外,在 2.3 ppm 附近可观察到 SP 的 7""" 和 8"""甲基信号峰[21],而化合物 II 在 2.3 ppm 附近则未见该信号,结合质谱分析初步证实化合物II 为 SP 的福洛胺糖上缺少两个甲基的产物。
在的 SP 生物合成基因簇中9 和22 是两个,-二甲基转移酶基因,两者有较高的同源性,负责催化福洛胺糖和麦洛胺糖上,-二甲基的形成。通过实验最终确定 Bsm22 蛋白负责催化形成福洛胺糖上的,-二甲基,而9很可能负责麦洛胺糖上的,-二甲基的催化,而且两者的功能不能互补。但没有分离出在麦洛胺糖上缺少二甲基的前体物,甚至没得到其他 SP 的前体物。其原因可能是麦洛胺糖是 SP 内酯环形成后连接的第一个脱氧糖,因为9基因阻断后,只形成了缺少,-二甲基麦洛胺糖,而这个底物不能被其糖基转移酶识别,导致后续的糖基化以及其他后修饰不能继续进行,无法形成完整的 SP,而内酯环不稳定易降解,因此未能得到其他 SP 的前体物。今后需要控制发酵时间来继续分离纯化 Δ9 变株中 SP 前体物。
多杀菌素 spinosyn 与蒽醌类抗生素 auricin 中也包含一个 D-福洛胺糖,[22]和52[23]是负责福洛胺糖的,-二甲基化的基因,Bsm9 与 SpnS 和 Sa52 蛋白序列的一致性分别为 47% 和 48%,Bsm22 蛋白序列与两者的一致性分别为 42% 和 68%,从同源性看,22 基因也更可能负责福洛胺糖的,-二甲基化。泰乐菌素上存在一个麦洛胺糖,其,-二甲基转移酶 TylM1 负责麦洛胺糖上的二甲基化并解析了其三维结构,阐明其催化的具体机制[24]。SP 基因簇中的 Bsm9 的蛋白序列与 TylM1 有较高的同源性,一致性达到 65%,而 Bsm22 与 TylM1 的一致性为 50%,从同源性上看 Bsm9 很可能是负责麦洛胺糖的,-二甲基化。另外在中的 DesVI 是另外一种,-二甲基转移酶,负责催化 dTDP-desosamine 上的-甲基化,其三维结构也已经被解析[25]。
在中 SP 生物合成基因簇中的两个,-二甲基转移酶基因9 和22 进行基因阻断后可以严重影响 SP 的合成及其抗菌活性,证实了两个,-二甲基是SP 抗菌活性的关键基团。福洛胺糖缺少二甲基的 SP 前体物,虽然抗菌活性下降,但 SP 还有其他抗炎和免疫调节活性,如果这个产物仍然保留这些生物学活性,将会有利于开发 SP 类似物的这些非抗菌活性而不必担心其产生抗生素的抗药性。缺失,-二甲基后的SP 前体物还可再进行化学修饰,可以得到一系列新的SP 衍生物,将为活性新化合物的筛选奠定基础。
[1] Harf R, Panteix G, Desnottes JF, et al. Spiramycin uptake by alveolar macrophages. J Antimicrob Chemother, 1988, 22 Suppl B:135-140.
[2] Kitzis M, Desnottes JF, Brunel D, et al. Spiramycin concentrations in lung tissue. J Antimicrob Chemother, 1988, 22 Suppl B:123-126.
[3] Smith CR. The spiramycin paradox. J Antimicrob Chemother, 1988, 22 Suppl B:141-144.
[4] Periti P, Mazzei T, Mini E, et al. Adverse effects of macrolide antibacterials. Drug Saf, 1993, 9(5):346-364.
[5] Amsden GW. Advanced-generation macrolides: tissue-directed antibiotics. Int J Antimicrob Agents, 2001, 18 Suppl 1:S11-S15.
[6] Wei HX, Wei SS, Lindsay DS, et al. A systematic review and meta-analysis of the efficacy of anti-toxoplasma gondii medicines in humans. PLoS One, 2015, 10(9):e0138204.
[7] Zeng S, Meng X, Huang Q, et al. Spiramycin and azithromycin, safe for administration to children, exert antiviral activity againstenterovirus A71 in vitro and in vivo. Int J Antimicrob Agents, 2019, 53(4):362-369.
[8] Pons S, Arrii E, Arnaud M, et al. Immunomodulation of endothelial cells induced by macrolide therapy in a model of septic stimulation. Immun Inflamm Dis, 2021, 9(4):1656-1669.
[9] Guangdong S, Jianlu D, Yiguang W. Construction and physiological studies on a stable bioengineered strain of shengjimycin. J Antibiot (Tokyo), 2001, 54(1):66-73.
[10] Thibessard A, Haas D, Gerbaud C, et al. Complete genome sequence of Streptomyces ambofaciens ATCC 23877, the spiramycin producer. J Biotechnol, 2015, 214:117-118.
[11] Karray F, Darbon E, Oestreicher N, et al. Organization of the biosynthetic gene cluster for the macrolide antibiotic spiramycin in Streptomyces ambofaciens. Microbiology (Reading), 2007, 153(Pt 12): 4111-4122.
[12] Karray F, Darbon E, Nguyen HC, et al. Regulation of the biosynthesis of the macrolide antibiotic spiramycin in Streptomyces ambofaciens.
J Bacteriol, 2010, 192(21):5813-5821.
[13] Choi SU, Kim MK, Ha HS, et al. In vivo functions of the gamma- butyrolactone autoregulator receptor in Streptomyces ambofaciens producing spiramycin. Biotechnol Lett, 2008, 30(5):891-897.
[14] Nguyen HC, Karray F, Lautru S, et al. Glycosylation steps during spiramycin biosynthesis in Streptomyces ambofaciens: involvement of three glycosyltransferases and their interplay with two auxiliary proteins. Antimicrob Agents Chemother, 2010, 54(7):2830-2839.
[15] Nguyen HC, Darbon E, Thai R, et al. Post-PKS tailoring steps of the spiramycin macrolactone ring in Streptomyces ambofaciens. Antimicrob Agents Chemother, 2013, 57(8):3836-3842.
[16] Yu QW. A new spiramycin-producing Streptomyces species. Acta Microbiol Sinica, 1982, 22(1):13-16. (in Chinese)
于其伟. 一株产生螺旋霉素的链霉菌新种. 微生物学报, 1982, 22(1):13-16.
[17] Nakahama K, Igarasi S. Microbial conversion of antibiotics. IV. Reduction of maridomycin. J Antibiot (Tokyo), 1974, 27(8):605-609.
[18] Nakahama K, Kishi T, Igarasi S. Microbial conversion of antibiotics. 3. Hydroxylation of maridomycin I and josamycin. J Antibiot (Tokyo), 1974, 27(6):433-441.
[19] Mo HB, Bai LQ, Wang SL, et al. Construction of efficient conjugal plasmids between Escherichia coli and Streptomycetes. Chin J Biotechnol, 2004, 20(5):662-666. (in Chinese)
莫宏波, 白林泉, 王胜兰, 等. 大肠杆菌-链霉菌高效接合载体的构建及其应用. 生物工程学报, 2004, 20(5):662-666.
[20] Huang H, Zheng G, Jiang W, et al. One-step high-efficiency CRISPR/Cas9-mediated genome editing in Streptomyces. Acta Biochim Biophys Sin (Shanghai), 2015, 47(4):231-243.
[21] Alam P, Barber J, Brennan RJ, et al. 1H and 13C NMR spectra of spiramycin I in organic and aqueous solutions. Magn Reson Chem, 1995, 33(3):228-231.
[22] Hong L, Zhao Z, Melançon CE 3rd, et al. In vitro characterization of the enzymes involved in TDP-D-forosamine biosynthesis in the spinosyn pathway of Saccharopolyspora spinosa. J Am Chem Soc, 2008, 130(14):4954-4967.
[23] Bekeova C, Rehakova A, Feckova L, et al. Characterisation of the genes involved in the biosynthesis and attachment of the aminodeoxysugar D-forosamine in the auricin gene cluster of Streptomyces aureofaciens CCM3239. Appl Microbiol Biotechnol, 2016, 100(7):3177-3195.
[24] Carney AE, Holden HM. Molecular architecture of TylM1 from Streptomyces fradiae: an N, N-dimethyltransferase involved in the production of dTDP-D-mycaminose. Biochemistry, 2011, 50(5):780-787.
[25] Burgie ES, Holden HM. Three-dimensional structure of DesVI from Streptomyces venezuelae: a sugar N,N-dimethyltransferase required for dTDP-desosamine biosynthesis. Biochemistry, 2008, 47(13):3982- 3988.
Identification of-dimethyltransferase gene function in spiramycin biosynthesis gene cluster
LIN Ru, LI Ke-meng, LIU Juan-juan, DAI Jian-lu, HE Wei-qing
Author Affiliation:CAMS Key Laboratory of Synthetic Biology for Drug Innovation, NHC Key Laboratory of Biotechnology of Antibiotics, Institute of Medicinal Biotechnology, Chinese Academy of Medical Sciences & Peking Union Medical College, Beijing 100050, China
To confirm the function of the two-dimethyltransferase genes9 and22 in the spiramycin (SP) biosynthetic pathway, and to isolate and purify the corresponding SP precursor from these gene disruption mutants.Antibiotic resistance gene insertion or CRISPR-Cas gene editing were used to9 and22 gene disruption and the gene complementation was also carried out in the mutants, respectively. Thin-layer chromatography (TLC) and high-performance liquid chromatography-mass spectrometry (HPLC-MS) were employed for analyze the fermentation products of the mutants Δ9 and Δ22. The demethylated SP precursors were purified by semi-preparative liquid chromatography, and its chemical structure was elucidated by nuclear magnetic resonance spectroscopy (NMR).Two gene disruption strains Δ9 and Δ22 were successfully constructed. The fermentation broth of the Δ9 mutant showed little antibacterial activity, and the corresponding SP precursors were not found by HPLC-MS analysis. However, the antibacterial activity of the fermentation product of the Δ22 decreased by more than 50%. The fermentation product isolated from the Δ22 was an SP precursor with forosamine devoid of,-dimethylation.The22 gene is responsible for the-dimethylation of forosamine in SP biosynthesis. The9 gene is probably involved in the,-dimethylation of mycaminose. No complementation was observed between the9 and22.
Spiramycin;,-dimethyltransferase gene; gene disruption
HE Wei-qing, Email: heweiqing@imb.pumc.edu
10.3969/j.issn.1673-713X.2022.04.002
国家自然科学基金(81773617、82073900);
中国医学科学院医学与健康科技创新工程(2021-1-I2M-1-028)
赫卫清,Email:heweiqing@imb.pumc.edu.cn
2021-11-16
猜你喜欢 二甲基基转移酶质粒 ——一道江苏高考题的奥秘解读和拓展">农杆菌转化法中的“Ti质粒转化载体系统”的简介——一道江苏高考题的奥秘解读和拓展中学生物学(2022年7期)2022-09-07全基因组测序后质粒的组装与鉴定研究进展*成都医学院学报(2022年4期)2022-08-19葡萄醇酰基转移酶编码基因遗传变异研究中国农业科学(2022年14期)2022-07-26氨基转移酶升高真有这么可怕吗保健与生活(2022年11期)2022-06-09菘蓝中咖啡酸氧甲基转移酶IiCOMT的克隆与表达分析中草药(2022年1期)2022-01-13氨基转移酶升高代表肝脏受损吗保健与生活(2021年5期)2021-04-12开发新方法追踪植物病害的全球传播(2020.6.7 iPlants)三农资讯半月报(2020年11期)2020-06-21小鼠转录因子STATl真核表达质粒的构建及生物学功能分析江苏农业学报(2019年1期)2019-09-10HCO3—对真空紫外/紫外/氯(VUV/UV/Cl)降解水中抗生素磺胺二甲基嘧啶的影响研究智富时代(2019年7期)2019-08-16HCO3—对真空紫外/紫外/氯(VUV/UV/Cl)降解水中抗生素磺胺二甲基嘧啶的影响研究智富时代(2019年7期)2019-08-16